Genotipificacion Del Virus de La Hepatitis C Por Secuenciacion
Diunggah oleh
Anonymous BD4x0u0 penilaian0% menganggap dokumen ini bermanfaat (0 suara)
48 tayangan1 halamanvhc
Hak Cipta
© © All Rights Reserved
Format Tersedia
PDF, TXT atau baca online dari Scribd
Bagikan dokumen Ini
Apakah menurut Anda dokumen ini bermanfaat?
Apakah konten ini tidak pantas?
Laporkan Dokumen Inivhc
Hak Cipta:
© All Rights Reserved
Format Tersedia
Unduh sebagai PDF, TXT atau baca online dari Scribd
0 penilaian0% menganggap dokumen ini bermanfaat (0 suara)
48 tayangan1 halamanGenotipificacion Del Virus de La Hepatitis C Por Secuenciacion
Diunggah oleh
Anonymous BD4x0uvhc
Hak Cipta:
© All Rights Reserved
Format Tersedia
Unduh sebagai PDF, TXT atau baca online dari Scribd
Anda di halaman 1dari 1
medigraphic
Artemisa
en lnea
DIFUSIN INTERNACIONAL
BM-5
GENOTIPIFICACIN DEL VIRUS DE LA HEPATITIS C POR SECUENCIACIN
DE LA REGIN 5UTR
Garca Lpez Eduardo Stanlin, Rocha Minerva Mata, Vzquez Oviedo Ramn Alberto, Carpermor, S.A. Alfonso Herrera No. 75, Col.
San Rafael, C.P. 06470, Mxico, D. F., E-mail: eduardo.garcia@carpermor.com.mx
Palabras clave: Genotipo, virus de la hepatitis C, Secuenciacin.
Introduccin: La hepatitis C es la forma ms comn de hepatitis post-transfunsional responsable de mayor al 70% de las
enfermedades virales del hgado, causada por el virus de la hepatitis C (VHC) que se transmite va parenteral y productos contaminados por sangre.1 El periodo de incubacin oscila entre 2 y
26 semanas.2 Pertenece a la familia Flavivirus, de un gnero separado: Hepacivirus. Es un virus envuelto con un tamao de 30 a
60 nm de dimetro, su genoma es RNA de cadena sencilla con
9,600 nucletidos, que codifica una protena que oscila entre
3,037-3,8008 aminocidos. 3 El VHC se ha estudiado
genticamente, siendo una de sus caractersticas la variabilidad
gentica: genotipos, subtipos y cuasiespecies. Esta variabilidad
gentica tiene importantes implicaciones tanto clnicas
(patogenicidad, diagnostico, resistencia al tratamiento) como
epidemiolgicas, de ah la importancia de la determinacin del
genotipo. El mtodo de referencia para la tipificacin molecular
del VHC es la secuenciacin completa del genoma viral y su
posterior anlisis filogentico.
Objetivo: Determinar el genotipo del VHC empleando tcnicas
moleculares, as como el diseo de primers, estandarizacin y
validacin del mtodo.
Metodologa: Diseos de primers: Se realiz el alineamiento de las
secuencias nucleotdicas de los diferentes genotipos del VHC
especficamente de la regin 5UTR (Regin no Traducible) con
el software BioEdit y el diseo de los primers para la RT-PCR y
la secuenciacin empleando el programa Primer3Plus; su sntesis se solicit con Applied Biosystems (AB). Extraccin de RNA
viral: Se realiz a partir del plasma de 10 pacientes, previamente
genotipificadas en Specialty Laboratories y de los genotipos de
ARMORED RNA con el kit QIAamp UltraSens Virus
(QIAGEN). RT-PCR y secuenciacin: Se amplific la regin
5UTR del VHC, se purific el producto de amplificacin a partir del gel de agarosa con el kit QIAquick Gel Extraction
(QIAGEN) y se realiz la reaccin de secuenciacin con el kit
BigDye Termninator v1.1. Cycle Sequencing (AB). La
secuenciacin fue llevada acabo por el equipo AB3130 Genetic
Analizer. Los resultados fueron alineados y analizados con el
Software DNA Sequencing Analysis v5.1 y SeqScape v2.5, tomando como secuencia de referencia la del genotipo 1a. Validacin de la prueba: Se realiz la sensibilidad, especificidad analtica,
linealidad de la prueba, precisin y exactitud.
Resultados: Con los primers diseados se obtuvo un amplificado de 270 pb y una secuenciacin de 240 pb. La especificidad
encontrada fue solo para VHC. En cuanto a la sensibilidad del
mtodo, limite de deteccin y/o cuantificacin fue de 500 UI/
mL, y la linealidad encontrada fue mayor o igual a ste valor.
Con respecto a la precisin, fue repetible y reproducible en un
100%; en exactitud, las secuencias determinadas estndares de
ARMORED RNA correspondieron a las reportadas por el
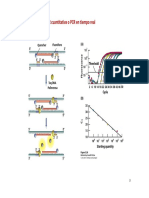
mismo de acuerdo a cada genotipo, de igual manera las 10 muestras analizadas correspondieron con lo reportado por el laboratorio de referencia, es decir en un 100%. La figura 1 muestra
algunas secuencias obtenidas junto con su genotipo, analizadas
con SeqScape v2.5.
Figura 1. Secuencias y electroferogramas que muestra uno de los
cambios de bases en los diferentes genotipos. (1) Secuencia nucleotdica
consenso; (2) Variantes de bases en cada genotipo; (3) Secuencia de
Referencia; (4) Aminocido codificado; 5) y (7) Secuencia
correspondiente al genotipo 1a; (6) y (8) Electroferograma del genotipo
1a; (9) Secuencia correspondiente al genotipo 2b; (10) Electroferograma
del genotipo 2b.
Discusin: Los resultados determinan que el mtodo amplifica
y secuencia especficamente VHC, de igual manera la sensibilidad, especificidad analtica, linealidad, precisin y exactitud son
resultados aceptables, lo que nos indican un mtodo especfico
y confiable para la determinacin del genotipo del VHC. La
figura 1 muestra la secuencia donde existe un cambio de base (C
por A) con respecto a la de referencia, lo que corresponde a un
genotipo 2b, sin embargo, cabe resaltar que este cambio es slo
uno de varios que son usados para tipificar un genotipo de otro.
Conclusiones: Se logr obtener un mtodo confiable y especiwww.medigraphic.com
fico para genotipificar el VHC por medio de secuenciacin de
62
ADN.
REFERENCIAS
1. Gmez CI, lvarez GM. Biologa y mtodos diagnsticos del virus
de la hepatitis C. Rev Biomed. 2003; 14: 253-68.
2. Robbins S, Kumar V, Cotran R. Robbins. Patologa humana. 7 ed.
Madrid: Elseiver; 2004.
3. Herreras JM, Diaz A, Jimnez M. Tratado de hepatologa. Sevilla:
Universidad de Sevilla; 1996.
MEMORIAS PROGRAMA
Anda mungkin juga menyukai
- Guía práctica para técnico superior de laboratorio de diagnóstico clínico y biomédicoDari EverandGuía práctica para técnico superior de laboratorio de diagnóstico clínico y biomédicoPenilaian: 4.5 dari 5 bintang4.5/5 (10)
- Jvizcaino, ABC Del Laboratorio Vol 25 No 2Dokumen4 halamanJvizcaino, ABC Del Laboratorio Vol 25 No 2Carolina PérezBelum ada peringkat
- PCR ModalidadesDokumen9 halamanPCR ModalidadesMarco OsunaBelum ada peringkat
- Diagnostico Molecular IIDokumen41 halamanDiagnostico Molecular IIMArgot FloresBelum ada peringkat
- Cuales Son Los Metodos Diagnosticos para Evidenciar El Material Genetico en Las BacteriasDokumen7 halamanCuales Son Los Metodos Diagnosticos para Evidenciar El Material Genetico en Las BacteriasMarianela Zeballos Merubia100% (1)
- Determinacion de La Carga Viral de Virus de Hepatitis C Mediante Cobas Taqman HCV Test v2Dokumen6 halamanDeterminacion de La Carga Viral de Virus de Hepatitis C Mediante Cobas Taqman HCV Test v2Hector MoragaBelum ada peringkat
- Copia de Examenes MolecularDokumen5 halamanCopia de Examenes MolecularSAUCEDO MANRIQUE VANESSA JACQUELINEBelum ada peringkat
- Infeccion Virus Inmunologia Felina Doble Reaccion en CadenaDokumen5 halamanInfeccion Virus Inmunologia Felina Doble Reaccion en CadenaCARLOTA DIAZ AGUILARBelum ada peringkat
- La Verdad Del VirusDokumen5 halamanLa Verdad Del VirusInt FibraBelum ada peringkat
- Biología Molecular en El Diagnóstico de Hepatitis ViralDokumen2 halamanBiología Molecular en El Diagnóstico de Hepatitis ViralJean Claudio Miranda ParraBelum ada peringkat
- Diagnóstico Coriza InfecciosaDokumen3 halamanDiagnóstico Coriza InfecciosaZolly Liz Isla CárdenasBelum ada peringkat
- Detección Del Provirus de Leucosis Bovina A Través de PCR AnidadaDokumen8 halamanDetección Del Provirus de Leucosis Bovina A Través de PCR AnidadaJose Salcedo ParedesBelum ada peringkat
- Trabajo de Biologia Molecular PCRDokumen4 halamanTrabajo de Biologia Molecular PCRMerlys ArvelaezBelum ada peringkat
- Reporte Escrito: Identificación de Infección Activa Del PatógenoDokumen11 halamanReporte Escrito: Identificación de Infección Activa Del PatógenoJuan Carlos Banda FonsecaBelum ada peringkat
- Sesión Modelo BioinfClásicaDokumen43 halamanSesión Modelo BioinfClásicaJuan José Vera AsaldeBelum ada peringkat
- A Qué Se Denominan Técnicas de Biología MolecularDokumen4 halamanA Qué Se Denominan Técnicas de Biología Molecularmarmergoz81Belum ada peringkat
- Genética - Semana 11Dokumen8 halamanGenética - Semana 11Jorge Zapata MenaBelum ada peringkat
- Examen Final Bio-Mol P1Dokumen5 halamanExamen Final Bio-Mol P1william chiluisaBelum ada peringkat
- Control de Calidad en Biologia MolecularDokumen65 halamanControl de Calidad en Biologia Molecular05-TL-HU-ERICK ANTONY BARZOLA GALDOSBelum ada peringkat
- Informme-Práctica - GenéticaDokumen8 halamanInformme-Práctica - GenéticaLUIS ALBERTO OTINIANO MENDEZBelum ada peringkat
- Cuestionario Equipo 1Dokumen5 halamanCuestionario Equipo 1Julio Amid Villegas CruzBelum ada peringkat
- Enfoque de QRT-PCR Optimizado para La Detección de ARN Intra y Extracelular Del SARS-CoV-2Dokumen5 halamanEnfoque de QRT-PCR Optimizado para La Detección de ARN Intra y Extracelular Del SARS-CoV-2Mariannis DiazBelum ada peringkat
- Trabajo Elisa InmunoDokumen7 halamanTrabajo Elisa InmunoAngelica TruyolBelum ada peringkat
- DX Molecular de Aspergilosis Pulmonar InvasivaDokumen24 halamanDX Molecular de Aspergilosis Pulmonar InvasivasisifoBelum ada peringkat
- PCR ConvencionalDokumen7 halamanPCR ConvencionalMARINA100% (5)
- Virus Influenza (H5N1) de Alta PatogenicidadDokumen3 halamanVirus Influenza (H5N1) de Alta PatogenicidadClaudio Zepeda del ValleBelum ada peringkat
- Development of RT-PCR For CoronavirusDokumen19 halamanDevelopment of RT-PCR For CoronavirusmalleakeylavBelum ada peringkat
- Semana 11 Práctica GenéticaDokumen9 halamanSemana 11 Práctica GenéticaAbdiel SuarezBelum ada peringkat
- Resumen: Detección Molecular y Tipificación Del Virus Dengue Por RT-PCR y PCR Anidada Usando Oligonucleótidos MejoradosDokumen6 halamanResumen: Detección Molecular y Tipificación Del Virus Dengue Por RT-PCR y PCR Anidada Usando Oligonucleótidos MejoradosRobert0GarcíaBelum ada peringkat
- Significación de Los Marcadores Infecciosos para Identificar Portadores de Hepatitis B en Donantes de SangreDokumen6 halamanSignificación de Los Marcadores Infecciosos para Identificar Portadores de Hepatitis B en Donantes de SangreAlin CelayaBelum ada peringkat
- Determinacion de La Carga Viral de Virus de Hepatitis B Mediante Cobas Taqman HBV TestDokumen6 halamanDeterminacion de La Carga Viral de Virus de Hepatitis B Mediante Cobas Taqman HBV TestHector MoragaBelum ada peringkat
- Guía Problemas 2C 2020Dokumen32 halamanGuía Problemas 2C 2020Camila RubinoBelum ada peringkat
- El Uso de Reacciones de PCR Anidadas Mejora La Tipificación Genética de Mycoplasma Hyopneumoniae A Partir de Diferentes Muestras ClínicasDokumen6 halamanEl Uso de Reacciones de PCR Anidadas Mejora La Tipificación Genética de Mycoplasma Hyopneumoniae A Partir de Diferentes Muestras ClínicasEdwin Aquino ApazaBelum ada peringkat
- S1 - Figs SuplementariasDokumen9 halamanS1 - Figs SuplementariasjnunevegBelum ada peringkat
- Importancia de Las Técnicas Moleculares en La Fisiología VegetalDokumen6 halamanImportancia de Las Técnicas Moleculares en La Fisiología VegetalBjerk PetersonBelum ada peringkat
- Trabajo Cibertorio - Designación Grupos e Instrucciones 202350Dokumen8 halamanTrabajo Cibertorio - Designación Grupos e Instrucciones 202350JENNIFER DANIELA ALQUINGA ANGOBelum ada peringkat
- Bioestadistica IIDokumen10 halamanBioestadistica IICarlos Silva OclochoBelum ada peringkat
- Informe Tecnicas Moleculares QPCRDokumen9 halamanInforme Tecnicas Moleculares QPCRCatalinaBelum ada peringkat
- Caracterizacion Antigenica Y Genetica para Determinar La Filogenia Del Virus de La Rabia en El Departamento de Puno en El PERIODO 1998 - 2002Dokumen20 halamanCaracterizacion Antigenica Y Genetica para Determinar La Filogenia Del Virus de La Rabia en El Departamento de Puno en El PERIODO 1998 - 2002JORGE PACHECOBelum ada peringkat
- ¿Qué Es?Dokumen5 halaman¿Qué Es?Sadaí ChacónBelum ada peringkat
- Meningitis RevisionDokumen18 halamanMeningitis Revisiongregory_housesBelum ada peringkat
- Obtención de Anticuerpos RecombinantesDokumen4 halamanObtención de Anticuerpos RecombinantesGUtsavoBelum ada peringkat
- Taller PCR en Tiempo Real FinalDokumen5 halamanTaller PCR en Tiempo Real FinalKaren HernándezBelum ada peringkat
- Resumen Microbiología 2Dokumen108 halamanResumen Microbiología 2marianatorres509Belum ada peringkat
- Desarrollo y Análisis Del Sistema Conjunto PCRDot BlotDokumen7 halamanDesarrollo y Análisis Del Sistema Conjunto PCRDot BlotMayra GameroBelum ada peringkat
- Hepatitis ViralesDokumen16 halamanHepatitis ViralesAnais Maria Fe PatiñoBelum ada peringkat
- Inmunología Dra Lucía Ortega Trabajo #8Dokumen11 halamanInmunología Dra Lucía Ortega Trabajo #8Nelson MezaBelum ada peringkat
- Intro - Metodologia FitoDokumen8 halamanIntro - Metodologia Fitodeneb denebBelum ada peringkat
- BTMAV INFORME Practicas Diseño de Primers para PCRDokumen20 halamanBTMAV INFORME Practicas Diseño de Primers para PCRAndrea Pignatelli100% (1)
- PCR, PCR Multiplex ExposicionDokumen32 halamanPCR, PCR Multiplex ExposicionGabby Alvarez Oviedo100% (1)
- PCR en Tiempo Real ExposicionDokumen19 halamanPCR en Tiempo Real ExposicionStefy TorresBelum ada peringkat
- Presentación 1Dokumen14 halamanPresentación 1Riquel LugoBelum ada peringkat
- Acidos Nucleicos y InmunocromatografíaDokumen6 halamanAcidos Nucleicos y InmunocromatografíaGaby Rojas pardoBelum ada peringkat
- Genetica TallerDokumen4 halamanGenetica TallerMaria AlejandraBelum ada peringkat
- PCR SalmonellaDokumen6 halamanPCR SalmonellaLusiana RodríguezBelum ada peringkat
- PCR MultiplexDokumen14 halamanPCR Multiplexoscarbio2009Belum ada peringkat
- Presentación de PowerPointDokumen33 halamanPresentación de PowerPointAna SalazarBelum ada peringkat
- El Virus de La Hepatitis CDokumen11 halamanEl Virus de La Hepatitis CChristian DominguezBelum ada peringkat
- Comparación de Ensayos de PCR en Tiempo Real para La Detección de Leptospira SPPDokumen10 halamanComparación de Ensayos de PCR en Tiempo Real para La Detección de Leptospira SPPJesús Enrique Turín VilcaBelum ada peringkat
- Artículo de Terapia Génica-SenbDokumen13 halamanArtículo de Terapia Génica-Senbsamuel.navarro5930Belum ada peringkat
- MicrosDokumen79 halamanMicrosRICHARD J BALVINBelum ada peringkat
- Examen BM SEP04Dokumen11 halamanExamen BM SEP04lord_minazaval3003Belum ada peringkat
- Guias de Tokio 2018Dokumen19 halamanGuias de Tokio 2018Andrés Escobar57% (7)
- Parcial 1Dokumen6 halamanParcial 1Griselda Marcos LinoBelum ada peringkat
- Grupos SanguíneosDokumen23 halamanGrupos SanguíneosTinkyWinkyBelum ada peringkat
- Hongos EntomopatogenosDokumen4 halamanHongos EntomopatogenosRicardoSagenBelum ada peringkat
- Examen Entrada MEDICINADokumen2 halamanExamen Entrada MEDICINAMaria Fernanda Delgado CisnerosBelum ada peringkat
- BacteriasDokumen155 halamanBacteriasIván ChapoñánBelum ada peringkat
- Enzimas UPAODokumen60 halamanEnzimas UPAOVictor Guevara ContrerasBelum ada peringkat
- Docsity Enfermedad de Chagas Malaria y LeishmaniosisDokumen15 halamanDocsity Enfermedad de Chagas Malaria y LeishmaniosisMaría Fernanda CamposBelum ada peringkat
- Investigación Producción de Vacunas-Andrea CedeñoDokumen25 halamanInvestigación Producción de Vacunas-Andrea CedeñoAndrea Cedeño LaidleyBelum ada peringkat
- Consenso Multidisciplinario Tratamiento Covid V8 PDFDokumen458 halamanConsenso Multidisciplinario Tratamiento Covid V8 PDFAlejandro Almeida MosqueraBelum ada peringkat
- 3.1 FISIOPATO DM-1 Dra V.TapiaDokumen18 halaman3.1 FISIOPATO DM-1 Dra V.TapiaDANIEL ALEJANDROBelum ada peringkat
- Medios de CultivoDokumen12 halamanMedios de CultivoLaila Nicol CalderonBelum ada peringkat
- HTLV-1 LeucemiaDokumen6 halamanHTLV-1 LeucemiaMyrcia Gaby Honorio Arroyo MendozaBelum ada peringkat
- Cuadro Diarreico AgudoDokumen33 halamanCuadro Diarreico AgudoCarlosFelipeBustillosSernaBelum ada peringkat
- DengueDokumen6 halamanDengueBrenda RosalesBelum ada peringkat
- Parainfluenza Canina, Maritza Hernández 3BDokumen17 halamanParainfluenza Canina, Maritza Hernández 3BMaritza Michelle Hernández CamachoBelum ada peringkat
- Prostaglandinas Y InterferonesDokumen4 halamanProstaglandinas Y InterferonesSulay HerreraBelum ada peringkat
- Resistencia A Los Antivirales de Acción Directa Contra El Virus de La Hepatitis CDokumen8 halamanResistencia A Los Antivirales de Acción Directa Contra El Virus de La Hepatitis CGustavo RuizBelum ada peringkat
- Cuadro Comparativo Procariotas y EucariotasDokumen1 halamanCuadro Comparativo Procariotas y EucariotasF CBelum ada peringkat
- BichosDokumen25 halamanBichosBryan GarcíaBelum ada peringkat
- Laboratorio N°9 Grupos SanguíneosDokumen11 halamanLaboratorio N°9 Grupos SanguíneosSun Hee RMBelum ada peringkat
- Aspartato AminotransferasaDokumen3 halamanAspartato AminotransferasaOmar XCBelum ada peringkat
- Taller Sample ENARMDokumen21 halamanTaller Sample ENARMLenny Camacho100% (2)
- Yaritza Anahi Hernandez GarciaDokumen4 halamanYaritza Anahi Hernandez GarciaJuan Alonso AltamiranoBelum ada peringkat
- PuchuDokumen9 halamanPuchuPilar PalaciosBelum ada peringkat
- Virología: Practica 03 - Titulación de VirusDokumen4 halamanVirología: Practica 03 - Titulación de VirusRomulo Aycachi Inga88% (8)
- VPHDokumen21 halamanVPHYessmi Ramos Miranda100% (2)
- Taller Recuperacion Grado DecimoDokumen1 halamanTaller Recuperacion Grado DecimoLeidi Milena Cristancho CruzBelum ada peringkat