Langkah Docking
Langkah Docking
Diunggah oleh
Rikzatus ZahrohJudul Asli
Hak Cipta
Format Tersedia
Bagikan dokumen Ini
Apakah menurut Anda dokumen ini bermanfaat?
Apakah konten ini tidak pantas?
Laporkan Dokumen IniHak Cipta:
Format Tersedia
Langkah Docking
Langkah Docking
Diunggah oleh
Rikzatus ZahrohHak Cipta:
Format Tersedia
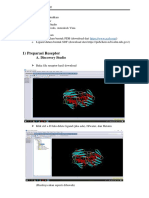
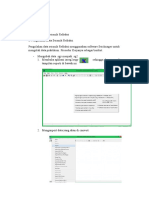
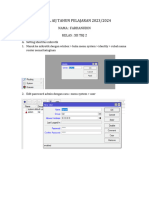
Buka aplikasi,masukkan 1xkk dengan mengeklik file >> impor molekul >> pilih 1xkk
2. Muncul tampilan, un centang water>> muncul gambar
3. Klik preparation >> detect cavities >> OK
4. Un centang kofaktor dan protein
5. Pilih cavities >> docking>> docking wizard
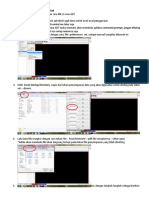
6. Refrence ligand >> pilih yang bawah (FMM_91 (A) >> next
7. Refrence ligand , pilih cavities yang sudah dipilih tadi >> klik next hingga muncul data output>>
mau disimpan dimana>> start
8. Skip run, lalu klik proses >> pilih RMSD yang paling kecil (di ss, dimasukin datanya) >> close
9. File >> import docking result
10. Pilih file yang tadi,akan muncul hasil data
11. Centang data dg RMSD paling kecil>> OK
12. Klik ligand map >> ligand (bagian atas) diganti dengan hasil data yang paling kecil tadi >> show
interaction
13. Kemudian di cari elemen elemennya, bisa di zoom in zoom out. Kemudian di ss
14. Buka chem draw >> convert name to structure >> masukkan nama obat >> OK >> struktur di copy
15. Buka chem draw 3D >> paste struktur
16. Klik file>>save as>>dikasih nama>> type di ubah menjadi “SYBYL2 (*mol2)>> save
17. Buka apk molegro>>file>> impor molekul>>pilih obat tadi>> open>>import
18. Klik docking >> docking wizard >>muncul tampilan, dan di centang obatnya (FMM_91 [A] di
uncentang) >> next >> pilih cavities yang sudah dipilih >> next terus sampai muncul data
output>>mau di simpan dimana>> OK>> Start >> continue >> skip run
19. Pencet proses >> Dilihat MolDock yang paling kecil >> close
20. Pencet file >>import docking result >> pilih yang kedua >> open
21. Centang MolDock yang paling kecil >> OK
22. Klik ligand map >> ligand (bagian atas) diganti dengan obatnya yang 00 >> show interaction
23. Buat tabel untuk semua data , dan ditambahkan hasil screenshot
Anda mungkin juga menyukai
- Faq DuktekDokumen39 halamanFaq DuktekSatya HanifBelum ada peringkat
- Langkah Molekular DockingDokumen2 halamanLangkah Molekular DockingMutmainnahBelum ada peringkat
- Modul Bioinfor DockingDokumen17 halamanModul Bioinfor DockinggitaBelum ada peringkat
- Tutorial Autodock VinaDokumen22 halamanTutorial Autodock VinapmuftiaBelum ada peringkat
- Langkah Chem DrawDokumen2 halamanLangkah Chem DrawSalsabila RaniahBelum ada peringkat
- Laporan Praktikum Molecular DockingDokumen14 halamanLaporan Praktikum Molecular DockingIsnaini100% (1)
- Cara Menginstall Minescape 4.1.1.8Dokumen13 halamanCara Menginstall Minescape 4.1.1.8Muhammad Tareq AdhaBelum ada peringkat
- Bioinformatika StepsDokumen8 halamanBioinformatika StepsAriane BeninaBelum ada peringkat
- Tutorial Autodock 4.2 - 2018Dokumen6 halamanTutorial Autodock 4.2 - 2018lokawati muliaBelum ada peringkat
- Kimia MedisinalDokumen19 halamanKimia MedisinalAnjali WidhiyaniBelum ada peringkat
- Progress Report Minggu ke-9Dokumen6 halamanProgress Report Minggu ke-9Masita AzzaBelum ada peringkat
- Logbook Ligan Alami Dan Reseptor Antikanker-DikonversiDokumen10 halamanLogbook Ligan Alami Dan Reseptor Antikanker-DikonversiNamira NaaziahBelum ada peringkat
- Tutorial Ukk Paket 2Dokumen18 halamanTutorial Ukk Paket 2NurhaeniBelum ada peringkat
- Prosedur KerjaDokumen11 halamanProsedur KerjaNurul MuhlisahBelum ada peringkat
- Tutorial Docking Autodock4.2 (New)Dokumen3 halamanTutorial Docking Autodock4.2 (New)MochamadFathurrBelum ada peringkat
- 3894 - Prosedur Chimera Dan VMDDokumen2 halaman3894 - Prosedur Chimera Dan VMDtirahmBelum ada peringkat
- Tutorial Docking Andi BasoDokumen7 halamanTutorial Docking Andi BasoHIDAYATBelum ada peringkat
- Install Minescape MincomDokumen2 halamanInstall Minescape Mincomdiamond_101190100% (1)
- Tutorial AutodockDokumen11 halamanTutorial AutodockA.S.H projecBelum ada peringkat
- Tutorial AutodockDokumen11 halamanTutorial AutodockFina RepitiyanaBelum ada peringkat
- (Update) PROSEDUR INSTALASI PATRAN NASTRAN 2012Dokumen4 halaman(Update) PROSEDUR INSTALASI PATRAN NASTRAN 2012Yang LekBelum ada peringkat
- (New) Membuat Manipulasi Data Pada Visual Basic 6Dokumen6 halaman(New) Membuat Manipulasi Data Pada Visual Basic 6helpdesk DanamonBelum ada peringkat
- DOCKINGDokumen8 halamanDOCKINGNovita ekaBelum ada peringkat
- LaporanDokumen13 halamanLaporanLia NurhayatiBelum ada peringkat
- Tugas 1 MikrokontrolerDokumen9 halamanTugas 1 MikrokontrolerMuhammad Adi RiswantoBelum ada peringkat
- DOCKING FEBYANTI SAFITRI Dan GUSTI MAHARINIDokumen7 halamanDOCKING FEBYANTI SAFITRI Dan GUSTI MAHARININasrullah HamdaniBelum ada peringkat
- Turorial Molecular DockingDokumen34 halamanTurorial Molecular DockinggedeBelum ada peringkat
- Alur DockingDokumen1 halamanAlur DockingAnnisa Zesika DewiBelum ada peringkat
- Laporan 1Dokumen30 halamanLaporan 1zhafira khairunnisa azraBelum ada peringkat
- MANUAL Cyclone 3DR - Volume TankiDokumen13 halamanMANUAL Cyclone 3DR - Volume TankiSurvey Geotech Lab SurabayaBelum ada peringkat
- Konfigurasi Debian 9 Semester 1Dokumen13 halamanKonfigurasi Debian 9 Semester 1Muhammad FahriBelum ada peringkat
- Ss KomdatDokumen12 halamanSs KomdatDevii KumalasarriBelum ada peringkat
- P.Komputer-Kls A - Tugas-01 - I MADE WIDHI ADITYA PRANATA - D021191073 - 2022Dokumen26 halamanP.Komputer-Kls A - Tugas-01 - I MADE WIDHI ADITYA PRANATA - D021191073 - 2022AdityaBelum ada peringkat
- Tutorial Pemetaan Arus Geostropik Menggunakan Surfer 13 Dan Odv 4Dokumen26 halamanTutorial Pemetaan Arus Geostropik Menggunakan Surfer 13 Dan Odv 4aldadestriantyBelum ada peringkat
- Panduan Setting MikrotikDokumen19 halamanPanduan Setting Mikrotikhendra akbarBelum ada peringkat
- Modul (Aij) Tahun Pelajaran 2023Dokumen16 halamanModul (Aij) Tahun Pelajaran 2023alsyaparelsimdigBelum ada peringkat
- Tutorial Docking AutodockDokumen3 halamanTutorial Docking AutodockFuzi Fauzia Fajrin Fauzia FajrinBelum ada peringkat
- Lembar Kerja 6Dokumen4 halamanLembar Kerja 6Chandra KusmayaBelum ada peringkat
- Cara Instal Minescape 4Dokumen2 halamanCara Instal Minescape 4Dhany PrasetyoBelum ada peringkat
- Langkah Kerja Composite Band ILWISDokumen5 halamanLangkah Kerja Composite Band ILWISakaHanAzfaBelum ada peringkat
- Modul (Aij) Tahun Pelajaran 2023Dokumen16 halamanModul (Aij) Tahun Pelajaran 2023alsyaparelsimdigBelum ada peringkat
- Modul 4 PPLL 2019 (Metodologi)Dokumen13 halamanModul 4 PPLL 2019 (Metodologi)Dedi NurpadilahBelum ada peringkat
- Skema Proses MD5Dokumen9 halamanSkema Proses MD5Muhammad Rachman NurhakimBelum ada peringkat
- Tutrial Tdyn by M. IqbalDokumen4 halamanTutrial Tdyn by M. IqbalAhmad FirdhausBelum ada peringkat
- Klasifikasi Otomatis Data LidarDokumen25 halamanKlasifikasi Otomatis Data LidarFajar Andi SugiyantoBelum ada peringkat
- Tugas PCD 3 - Kholifatussholikhakh - 21110118120023Dokumen38 halamanTugas PCD 3 - Kholifatussholikhakh - 21110118120023Caramel LatteBelum ada peringkat
- Panduan Molecular Docking Autodock 4.0Dokumen17 halamanPanduan Molecular Docking Autodock 4.0alisia difianaBelum ada peringkat
- Log Book Preparasi Ligan Dan Reseptor - Kelompok Antihistamin - Farmasi D 2019Dokumen10 halamanLog Book Preparasi Ligan Dan Reseptor - Kelompok Antihistamin - Farmasi D 2019Namira NaaziahBelum ada peringkat
- Export Logfile DT Kedalam MapinfoDokumen10 halamanExport Logfile DT Kedalam MapinfoJanuar LengobzBelum ada peringkat
- LA VmwareDokumen27 halamanLA VmwareWidho Awan FirnandoBelum ada peringkat
- Pengenalan R Dan SASDokumen8 halamanPengenalan R Dan SASReza MuhammadiBelum ada peringkat
- Panduan Migrasi Fingerspot - iO-Tool MigrasiDokumen12 halamanPanduan Migrasi Fingerspot - iO-Tool MigrasiInnalillaahi Wa inna ilaihi raaji'uunBelum ada peringkat
- Manual Book Pt. Pupuk Kujang - Software Truck Scale PDFDokumen35 halamanManual Book Pt. Pupuk Kujang - Software Truck Scale PDFAdam Rahmat FajarBelum ada peringkat
- Faq Seal Offline Job 2Dokumen3 halamanFaq Seal Offline Job 2KumanBelum ada peringkat
- Faq Seal Offline Job 2Dokumen3 halamanFaq Seal Offline Job 2Amril-kunBelum ada peringkat
- Validasi ReseptorDokumen3 halamanValidasi Reseptorsyifa nurul ainiBelum ada peringkat