Laporan
Diunggah oleh
Lia NurhayatiJudul Asli
Hak Cipta
Format Tersedia
Bagikan dokumen Ini
Apakah menurut Anda dokumen ini bermanfaat?
Apakah konten ini tidak pantas?
Laporkan Dokumen IniHak Cipta:
Format Tersedia
Laporan
Diunggah oleh
Lia NurhayatiHak Cipta:
Format Tersedia
Laporan Praktikum Kimia Komputasi Modul 5
I. Tujuan
1. Melakukan persiapan untuk molecular docking
2. Melakukan validasi metode docking
3. Melakukan analisis hasil molecular docking
II. Prinsip
Teknik penambatan ligand ke dalam sisi aktif reseptor yang dilanjutkan dengan
evaluasi molekul berdasarkan kecocokan konformasi struktur dan sifat.
III. Teori Dasar
Simulasi molecular docking merupakan suatu simulasi yang dapat digunakan untuk
mempelajari interaksi antara ligan dengan protein atau interaksi antar protein pada
level atom. Tujuan studi docking adalah untuk mendapatkan pasangan terbaik dan
memperoleh informasi konformasi dan afinitas.
Interaksi yang terjadi apabila suatu ligan ditempelkan pada suatu makromolekul,
akan mempengaruhi struktur 3 dimensinya, yang dipengaruhi oleh adanya kontribusi
interaksi elektrostatik, van der waals, ikatan hidrogen dan sumbangan interaksi yang
lain. Akibatnya ada beberapa kemungkinan tipe konformasi bersama yang terbentuk.
Konformasi yang terbentuk tersebut dalam docking diketahui dengan melihat tempat
penempelan (binding site), tipe penempelan (binding mode) dan energi penempelan
(binding energi) (Aatukaapru & Janne ojanen, 2002)
Menurut Kitchen et al., 2004 ada dua masalah umum yang dijumpai dalam
docking molekul, masalah yang pertama adalah pemilihan fungsi energi yang
digunakan selama proses docking serta pemilihan algoritma yang tepat. Autodock
merupakan software yang digunakan untuk proses docking. Algoritma yang disediakan
dalam proses docking pada autodock adalah SA (Simulated Annealing Monte Carlo),
algoritma genetik, Local search (LS) dan algoritma Lamarckian Genetik. Pemetaan
dengan algoritma SA dilakukan secara acak ke seluruh area pada protein. Kelemahan
metode ini hanya cocok untuk ligan yang fleksibelitasnya maksimal 8 ikatan yang
mampu berotasi, diatas 8 perhitungan akan menjadi sulit karena banyaknya
1 Dini Anggraeni 11141112
kemungkinan konfigurasi dalam docking sehingga global minimal sulit didapatkan.
Algoritma Genetik dikenal juga sebagai traditional Darwinian genetic algoritm,
karena dasar yang digunakan adalah teori genetik dan evolusi Darwin. Proses
pemetaan tidak dilakukan secara acak sehingga peluang global minimal tercapai cukup
besar, namun untuk protein yang memiliki cekungan (cavity) yang cukup dalam
langkah yang dilakukan banyak sehingga kurang efisien untuk mendapatkan posisi
minimal. Local Search (LS), LS merupakan algoritma mencari energi pada lokal atau
cekungan tertentu . LGA (Lamarckian genetic Algoritm) merupakan algoritma
gabungan antara GA-LS (Genetic algoritma-local search), disebut LGA karena anak
diperbolehkan untuk mewarisi adaptasi local search dari orang tuanya. Pada LGA
optimasi global dilakukan mengunakan GA sedang optimasi lokal digunakan LS.
Dengan algoritma gabungan ini cekungan yang cukup dalam yang dilakukan
perhitungan sedang cekungan yang lain tidak terlalu diperhitungkan, kelebihan metode
ini proses docking menjadi lebih efisien dan langkah yang digunakan tidak terlalu
panjang (Huey and Morries., 2003).
Tahapan Dalam Simulasi Docking:
1.Persiapan file input struktur protein yang sebelumnya telah dipreparasi
2.Persiapan file input struktur ligan
3.Validasi docking
4.Docking ligan uji menggunakan parameter hasil validasi
5.Analisis Hasil Docking
IV. Prosedur
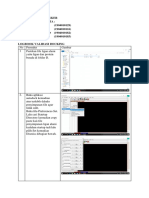
1. Buka file .pdb dari 4AC2 di discovery studio visualizer 2016
2 Dini Anggraeni 11141112
2. Buka hierarchy untuk melihat komponen yang ada pada file .pdb ini. Caranya
klik view> hierarchy > (beri tanda centang paa hierarchy)
3. Hilangkan komponen air, kemudian pisahkan 43F dari protein. Simpan file
protein (masih mengandung etilen glikol dan NAD) dengan format .pdb (simpan
dengan nama 4AC2-protein.pdb).
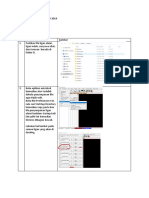
4. Kemudian buka lagi file 4AC2.pdb yang masih utuh. Hilangkan semua
komponen kecuali 43F. Simpan file 43F format sybyl mol2 ( simpan dengan
nama 43F.mol2)
5. Buka perangkat lunak autodock tools
Berikut adalah tampilan autodock tools
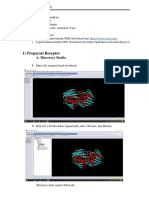
6. Buka file protein dengan cara file > read molecule, kemudian pilih file 4AC2-
protein.pdb.
7. File *.pdb ini tidak memiliki atom hidrogen, maka sebelum melakukan simulasi,
atom hidrogen harus ditambahkan terlebih dahulu. Klik edit > hidrogen > add.
Kemudian akan muncul dialog box seperti berikut Pilih polar only kemudian ok.
Amati perubahan yang terjadi pada visualisasi protein.
8. Simpan file PDB protein yang telah ditambahkan hidrogen. Caranya file > save
> writePDB. Simpan protein pada folder tempat instalasi autodock tools.
9. Sembunyikan tampilan file protein, kemudian masukan file 43F.
3 Dini Anggraeni 11141112
Klik kanan pada tulisan 4AC2_protein, kemudian hide molecule. Untuk
memasukan ligan, klik ligand > input > open. Pilih file 43F.mol2.
10. Klik ligand > torsion tree > detect root
11. Klik ligand > torsion tree > choose torsions. Pastikan jumlah ikatan yang dapat
berotasi maksimum, namun tidak melebihi 32
12. Klik ligand > output > save PDBQT
Simpan file ligan dengan format .pdbqt di folder tempat autodock tools.
13. Sebelum melakukan run docking, harus dilakukan run autogrid terlebih dahulu.
Persiapan file input autogrid adalah sebagai berikut:
Grid > macromolecule > choose
Pilih file protein > choose. Tunggu dan kemudian akan diminta untuk
menyimpan file protein dalam format .pdbqt. simpan file protein
tersebut kedalam format .pdbqt difolder tempat instalasi autodock tools
Grid > set map types > choose ligand
Pilih 43F, kemudian choose.
Grid > grid box
Untuk validasi pertama, pilih center > center on ligand. Ukuran box dapat
disesuaikan jika ligand ujinya besar. Pada contoh kali ini, cukup gunakan
box 40 x 40 x 40 jika sudah selesai mengatur, klik file > close saving
current.
Grid > output > save GPF
Simpan file .gpf pada folder tempat instalasi autodock tools. Biasanya,
.gpf harus diketik secara maual karena tidak secara otomatis terketik. Pada
contoh kali ini, beri nama 4AC2-validasi.gpf
14. Run > run autogrid
Pada bagian program pathname, pilih browse dan cari file aplikasi autogrid
(autogrid.exe). file aplikasi ini akan berada pada folder system32 ditempat
4 Dini Anggraeni 11141112
instalasi. Kemudian pada bagian parameter file, pilih browse dan pilih file .gpf
yang tadi sudah disiapkan, maka setelah memilih file .gpf, secara otomatis akan
muncul log filename. Dibagian cmd, pastikan bahwa setelah nama file .gpf, ada
tanda -|, kemudian berikutnya langsung nama log nya. Hapus tulisan path yang
ada didepannya (lihat pada contoh gambar diatas).
Klik launch.
Akan muncul dialog box seperti gambar diatas. Hal ini menandakan proses
docking sedang berlangsung. Tunggu hingga dialog box ini menghilang secara
otomatis, yang artinya proses telah selesai dilakukan.
Jika proses sudah selesai, buka bagian kotak hitam autodock tools. Cek apakah
proses berhasil atau tidak.
15. Setelah proses autogrid ini selesai dilakukan, lakukan persiapan untuk
melakukan proses docking. Persiapan file inputnya adalah sebagai berikut:
a. Docking > Macromolecule > Set Rigid Filename
Kemudian pilih file 4AC2_protein.pdbqt.Klik open
b. Docking > ligand > choose
5 Dini Anggraeni 11141112
Kemudian pilih 43F > Select ligand > accept
c. Docking > Search Parameter > Genetic Algorithm
Pada bagian Number of GA Runs, diisi dengan berapa banyak konformasi
pencarian yang diinginkan. Kemudian pilih Accept setelah selesai mengatur
parameter
d. Docking>Output> Lamarckian GA.
Simpan file *.dpf pada folder tempat instalasi autodock Tools. Ketik *.dpf secra
manual, beri nama 5ANG-validasi.dpf
16. Run > Run autodock.
Pengaturan pada tahap ini sama dengan Autogrid pada tahap nomor 15. Hanya
saja ganti file autogrid.exe dengan autodock4.exe. kemudian file *.gpf pada
autogrid, diganti dengan *.dpf pada saat running autodock sehingga file output
berupa file *.dlg
Klik launch dan tunggu hingga proses selesai
6 Dini Anggraeni 11141112
17. Setelah selesai, lakukan analisis terhadap hasil redocking. Cek apakah nilai
RMSD < 2 A
18. Jika hasil RMSD tidak <2 A berarti parameter docking tidak valid. Ulangi
proses redocking dengan pengaturan parameter yang lain. jika hasil RMSD <2A,
parameter docking sudah valid.
19. Jika parameter docking valid, catat pengaturan gridbox yang digunakan pada
saat proses redocking.
Cari tampilan seperti berikut pada file 5ANG-validasi.dlg di notepad.
Dari data diatas, dapat diketahui bahwa ukuran gridbox yang digunakan asdalah
40 x 40 x 40 untuk spacing 0,375 A. Coordinate central grid point = (-1.825,
7.588, 14.248)
V. Data Percobaan
4AC2 (CRYSTAL STRUCTURE OF TRANSTHYRETIN IN COMPLEX WITH
LIGAND C-7
7 Dini Anggraeni 11141112
VI. Hasil
Protein yang telah dipisahkan dengan ligan alaminya menggunakan aplikasi Discovery
Studio
Protein 4AC2 Ligan 43F dari protein 4AC2
8 Dini Anggraeni 11141112
Pengecekan Torsion Tree pada Ligan 43F
Lebih banyak area berwarna merah dibandingkan area berwarna hijau. Dimana artinya
struktur tersebut tidak dapat diputar, dan jika tidak dapat berputar maka senyawa
tersebut dikatakan rigid.
Ukuran Grid Box untuk validasi dan Visualisasi Grid Box pada validasi
9 Dini Anggraeni 11141112
Tampilan pada saat melakukan autogrid pada ligan 43F
10 Dini Anggraeni 11141112
Run autogrid sukses karena pada saat akan menjalankan autogrid tidak ada yang
salah sehingga run successful
Pada saat melakukan parameter docking, untuk number of GA Runs dipilih nomor
10, dan untuk maximum number of evals dipilih medium
11 Dini Anggraeni 11141112
Nilai RMSD dari hasil re-docking antara ligan alami 43F dengan proteinnya 4AC2
memiliki nilai RMSD dibawah 2A, sehingga dapat dikatakan valid.
Pengaturan Grid box.
Dari hasil validasi didapat nilai Grid maps will be centered on user-defined
coordinate (0,204, -0,194, 15,961) sehingga gridbox ligan disesuaikan dengan
koordinat tersebut
12 Dini Anggraeni 11141112
VII. Kesimpulan
Dari hasil analisis docking yaitu nilai RMSD pada protein 4AC2 dengan ligand 43F
menunjukkan hasil yang valid karena nilai dari re-docking tersebut dibawah 2A.
VIII. Daftar Pustaka
1. http://www.rcsb.org/pdb/explore/explore.do?structureId=4AC2
2. Morris, G.M., Goodsell. D. S., Halliday,R.S., Huey, R. Hart. W.E, et al., 1998,
Automated Docking Using Lamarckian Genetic Alghorithmand an Empirical
Binding free Energy function, J Comput Chem.
13 Dini Anggraeni 11141112
Anda mungkin juga menyukai
- Laporan Praktikum Kimia KomputasiDokumen19 halamanLaporan Praktikum Kimia KomputasiSHELFI RENITABelum ada peringkat
- Turorial Molecular DockingDokumen34 halamanTurorial Molecular DockinggedeBelum ada peringkat
- Panduan Molecular Docking Autodock 4.0Dokumen17 halamanPanduan Molecular Docking Autodock 4.0alisia difianaBelum ada peringkat
- Modul Biokimia Proyek 2: Molecular DockingDokumen31 halamanModul Biokimia Proyek 2: Molecular DockingNISRINA NAJLA HUWAIDABelum ada peringkat
- Tutorial AutodockDokumen11 halamanTutorial AutodockFina RepitiyanaBelum ada peringkat
- Tutorial AutodockDokumen11 halamanTutorial AutodockA.S.H projecBelum ada peringkat
- Kimia Komputasi Modul 5Dokumen23 halamanKimia Komputasi Modul 5rillynBelum ada peringkat
- Mari Belajar Molecular DockingDokumen5 halamanMari Belajar Molecular DockingAnggie WindrianiBelum ada peringkat
- LOGBOOK KELOMPOK ANTIKANKER VALIDASI DOCKING-dikonversiDokumen12 halamanLOGBOOK KELOMPOK ANTIKANKER VALIDASI DOCKING-dikonversiNamira NaaziahBelum ada peringkat
- Kimed 6Dokumen7 halamanKimed 6rizaldi rahmatullahBelum ada peringkat
- Laporan Kimkom Modul 5 Fix Alsa Sah AlhamdulilahDokumen19 halamanLaporan Kimkom Modul 5 Fix Alsa Sah AlhamdulilahAlsa giani MaheshaBelum ada peringkat
- Bioinformatika StepsDokumen8 halamanBioinformatika StepsAriane BeninaBelum ada peringkat
- Tutorial Autodock 4.2 - 2018Dokumen6 halamanTutorial Autodock 4.2 - 2018lokawati muliaBelum ada peringkat
- Logbook Docking Senyawa Ligan Alami, Ligan Induk, Ligan Turunan Dan Senyawa Obat - Kelampok 6 Farmasi D 2019Dokumen10 halamanLogbook Docking Senyawa Ligan Alami, Ligan Induk, Ligan Turunan Dan Senyawa Obat - Kelampok 6 Farmasi D 2019Elis Nurul IkhlasBelum ada peringkat
- B2 1 1908010096 Khairunnisa Lapres p2Dokumen14 halamanB2 1 1908010096 Khairunnisa Lapres p2Khairun nisaBelum ada peringkat
- Tutorial Docking AutodockDokumen3 halamanTutorial Docking AutodockFuzi Fauzia Fajrin Fauzia FajrinBelum ada peringkat
- Tutorial Molecular Docking Dengan Autodock VinaDokumen7 halamanTutorial Molecular Docking Dengan Autodock Vinaludya pulungBelum ada peringkat
- Molecular DockingDokumen6 halamanMolecular DockingYasaKaryadaBelum ada peringkat
- Logbook Kimed IiDokumen11 halamanLogbook Kimed IiTatang SupriatnaBelum ada peringkat
- Tutorial Docking Autodock4.2 (New)Dokumen3 halamanTutorial Docking Autodock4.2 (New)MochamadFathurrBelum ada peringkat
- Tutorial Autodock VinaDokumen22 halamanTutorial Autodock VinapmuftiaBelum ada peringkat
- Laporan Praktikum Molecular DockingDokumen14 halamanLaporan Praktikum Molecular DockingIsnainiBelum ada peringkat
- Cara Docking Senyawa Dengan Autodock 4Dokumen10 halamanCara Docking Senyawa Dengan Autodock 4Carlos RumangunBelum ada peringkat
- DOCKINGDokumen8 halamanDOCKINGNovita ekaBelum ada peringkat
- Tutorial Docking Andi BasoDokumen7 halamanTutorial Docking Andi BasoHIDAYATBelum ada peringkat
- Modul Sendiri LolDokumen33 halamanModul Sendiri LolyaminBelum ada peringkat
- 11-BRIYAN NUGRAHA PUTRA-06-Praktek Pencarian Dan Pengurutan DataDokumen16 halaman11-BRIYAN NUGRAHA PUTRA-06-Praktek Pencarian Dan Pengurutan DataGofur SincanBelum ada peringkat
- Carakerja TyyDokumen2 halamanCarakerja TyyEfanBelum ada peringkat
- BAB IV - Nurul AeniDokumen4 halamanBAB IV - Nurul AeniKudus Abdul azizBelum ada peringkat
- Modul Bioinfor DockingDokumen17 halamanModul Bioinfor DockinggitaBelum ada peringkat
- 06-ALFANDI PRIYATNA-06-Praktek Pencarian Dan Pengurutan DataDokumen15 halaman06-ALFANDI PRIYATNA-06-Praktek Pencarian Dan Pengurutan DataGofur SincanBelum ada peringkat
- Bioinformatika 4Dokumen9 halamanBioinformatika 4pwd bambangBelum ada peringkat
- Tutorial Simulasi Molecular Docking Menggunakan DOCK6Dokumen6 halamanTutorial Simulasi Molecular Docking Menggunakan DOCK6putryaBelum ada peringkat
- Additional ModulDokumen14 halamanAdditional ModulYudi PratamaBelum ada peringkat
- Contoh AplikasiDokumen17 halamanContoh AplikasiAiguille DevoirBelum ada peringkat
- Validasi ReseptorDokumen3 halamanValidasi Reseptorsyifa nurul ainiBelum ada peringkat
- Modul 7 (Penambatan Molekul)Dokumen2 halamanModul 7 (Penambatan Molekul)Ruth AnnekeBelum ada peringkat
- Protokol Pelatihan BioinformatikaDokumen21 halamanProtokol Pelatihan BioinformatikaI EBelum ada peringkat
- B2 - 3 - 1908010108 - Muhammad Naufal Rabbani - P2Dokumen14 halamanB2 - 3 - 1908010108 - Muhammad Naufal Rabbani - P2siti salma haniyyahBelum ada peringkat
- Cinta Skripsi Banget-46-54Dokumen9 halamanCinta Skripsi Banget-46-54Fyan VergarraBelum ada peringkat
- Laporan Praktikum Modul 2 Sistem DigitalDokumen8 halamanLaporan Praktikum Modul 2 Sistem Digitalkyrgios50% (2)
- Laporan Praktikum Kimia Komputasi Virtual Screening-DikonversiDokumen17 halamanLaporan Praktikum Kimia Komputasi Virtual Screening-DikonversiTiktok SerBelum ada peringkat
- DOCKING FEBYANTI SAFITRI Dan GUSTI MAHARINIDokumen7 halamanDOCKING FEBYANTI SAFITRI Dan GUSTI MAHARININasrullah HamdaniBelum ada peringkat
- Laporan Praktikum DockingDokumen14 halamanLaporan Praktikum DockingMuhammadRezaPahleviBelum ada peringkat
- Acara 3. Desain PrimerDokumen10 halamanAcara 3. Desain PrimerSalsabila Fitra RamadantiBelum ada peringkat
- Septiani Nurcahyanti - 24020221140090 - Lapres Acara 4 BioinformatikaDokumen14 halamanSeptiani Nurcahyanti - 24020221140090 - Lapres Acara 4 BioinformatikaSeptiani NurcahyantiBelum ada peringkat
- Trie Oktaviani - Penambatan Molekul (Molecular Docking)Dokumen17 halamanTrie Oktaviani - Penambatan Molekul (Molecular Docking)Trie OktavianiBelum ada peringkat
- Bab 3Dokumen6 halamanBab 3sukmawati Farm017Belum ada peringkat
- Combinational Logic SequenceDokumen9 halamanCombinational Logic SequenceYogi Salomo Mangontang PratamaBelum ada peringkat
- Perencanaan Transportasi Operatif - Solusi Dalam Rantai Pasokan Barang Konsumen-Physica-1-1Dokumen40 halamanPerencanaan Transportasi Operatif - Solusi Dalam Rantai Pasokan Barang Konsumen-Physica-1-1La Ode Agustian NurrahmanBelum ada peringkat
- Cake Rna RasiyyDokumen5 halamanCake Rna RasiyyEfanBelum ada peringkat
- Kimkim HendrikDokumen11 halamanKimkim HendrikHendrik SeptiadiBelum ada peringkat
- Resume 6Dokumen9 halamanResume 6004Nelly JuentiBelum ada peringkat
- Pre Test TaqiyuddinDokumen3 halamanPre Test TaqiyuddinAhmad TaqiyuddinBelum ada peringkat
- Nelly Juenti - Resume 6 - Kelompok 2Dokumen9 halamanNelly Juenti - Resume 6 - Kelompok 2004Nelly JuentiBelum ada peringkat
- Pengolahan Dinsar Dengan Software Snap-1Dokumen27 halamanPengolahan Dinsar Dengan Software Snap-1An Nisa Tri Rahmawati50% (2)
- Qiime2 ProtocolDokumen11 halamanQiime2 ProtocolfarrassBelum ada peringkat
- Object Detection Tensorflow API Part 2 Data PreparationDokumen4 halamanObject Detection Tensorflow API Part 2 Data PreparationBaiqunyBelum ada peringkat
- Molecular DockingDokumen57 halamanMolecular DockingpritaultimardianBelum ada peringkat
- Filsafat Sejarah-1Dokumen4 halamanFilsafat Sejarah-1Lia NurhayatiBelum ada peringkat
- Sejarah Pergerakan Nasional (Jaman Jepang)Dokumen27 halamanSejarah Pergerakan Nasional (Jaman Jepang)Lia NurhayatiBelum ada peringkat
- ProdiaDokumen3 halamanProdiaLia NurhayatiBelum ada peringkat
- Definisi IDokumen9 halamanDefinisi ILia NurhayatiBelum ada peringkat