Hasil Materi 4
Diunggah oleh
nur aisyah adzuti0 penilaian0% menganggap dokumen ini bermanfaat (0 suara)
4 tayangan5 halamanJudul Asli
hasil materi 4
Hak Cipta
© © All Rights Reserved
Format Tersedia
DOCX, PDF, TXT atau baca online dari Scribd
Bagikan dokumen Ini
Apakah menurut Anda dokumen ini bermanfaat?
Apakah konten ini tidak pantas?
Laporkan Dokumen IniHak Cipta:
© All Rights Reserved
Format Tersedia
Unduh sebagai DOCX, PDF, TXT atau baca online dari Scribd
0 penilaian0% menganggap dokumen ini bermanfaat (0 suara)
4 tayangan5 halamanHasil Materi 4
Diunggah oleh
nur aisyah adzutiHak Cipta:
© All Rights Reserved
Format Tersedia
Unduh sebagai DOCX, PDF, TXT atau baca online dari Scribd
Anda di halaman 1dari 5
IV.
Cara Kerja
4.1. Waktu dan Tempat
Praktikum dilaksanakan pada hari Rabu, 15 Maret 2023 pada pukul 10.00
sampai dengan 12.00 WIB. Praktikum dilaksanakan di Laboratorium
Mikrobiologi Jurusan Biologi Fakultas Matematika dan Ilmu Pengetahuan Alam
Universitas Sriwijaya, Indralaya.
4.2. Alat dan Bahan
Alat yang digunakan pada praktikum berupa laptop, GeneStudio, java,
mesquite, dan mega 11. Bahan yang digunakan berupa data DNA hasil
sekuensing.
4.3. Cara Kerja
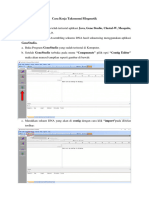
Aplikasi Gene Studio dilakukan dengan membuka programnya, lalu pilih
menu “Components” dan “Contig Editor”. Dimasukkan sekuen DNA yang di
contig dengan cara klik “import” pada deretan toolbar. Kemudian pilih file hasil
sekuensing yang akan diedit baik file forward maupun reverse dengan eksistensi
format file AB1. Diblok kedua file kemudian diklik Open. Selanjutnya, klik
“Open” maka program secara otomatis akan assemble guna digabungkan kedua
file menjadi satu bagian. Sebelum file tampil, akan muncul kotak dialog opsi
mengatur tingkat toleransi pemasangan nukleotida (semakin tinggi yang dipilih,
maka program hanya akan mencocokan pasangan nukleotida dengan nilai tinggi),
klik OK. Jika grafik romato gram tidak otomatis keluar, pilih dan klik “Show all”.
Sekuens forward memiliki tanda panah arah kekanan, sedangkan reverse
arah kekiri, jika tanda panah sekuens forward tidak mengarah kekanan atau tidak
sesuai maka lakukan “Reverse Complement” melalui toolbar “Contig” kemudian
klik “Reverse”. Cek kembali arah panah, jika benar lanjut lakukan koreksi pada
basa nukleotida yang masih bermasalah atau eror dengan fungsi “Ambiguity”.
Dipilih toolbar “Contig” lalu “Ambiguity Finder” dan akan terdapat dua opsi
“Before ambiguity” kursor akan berada di Universitas Sriwijaya belakang
nukleotida yang salah. Setelah seluruh nukleotida benar, bias dilakukan export
untuk dikonversi dalam bentuk FASTA, pilih “export consensus” pada toolbar
berinama file dan simpan dengan eksistensi file format FASTA klik Ok atau Save.
Setelah proses assembling sekuen DNA hasil sekuensing selesai langkah
selanjutnya yaitu Homologi Search menggunakan menu Basic Local Alignment
Search Tool (BLAST) pada NCBI. Untuk sampel DNA pilih “Nucleotide
BLAST”. Dimasukkan file FASTA (.fas) pada note pad dengan cara copy paste,
atau pilih browser pada bagian bawah dan open file dimana tempat kita
menyimpan data sampel hasil pembacaan dengan Gene Studio. Apabila semua
kotak isi antelah sesuai, lalu Run BLAST. Terdapat 100 data (scroll up dan scroll
down). Tahap selanjutnya, dipilih 9 spesies yang berbeda den gancar ater lebih
dahulu klik select all pada pojok kiri atas, lalu centang 9 spesies bakteri yang
diinginkan, setelah itu klik opsi download dan pilih FASTA (Complete
Sequence).
Masuk ke mesquite dahulu disatukan file hasil download BLAST dengan
file hasil assembling DNA pada Gene Studio dengan membuka kedua file lalu
copy sekuens hasil assembling dan tekan pada bagian paling atas pada file
download pada laman NCBI dan setelah disatukan lalu save as file tersebut diberi
nama file “Hasil Homologi Search Sismik 1” dan rubah ekstensi file nya dengan
cara tambah .nt tanpa spasi dibelakang nama file. Kemudian buka Mesquite lalu
klik File dan pilih Open file serta pilih file yang telah dibuat pada langkah
sebelumnya, kemudian klik Open. Dipilih FASTA (DNA/RNA) sesuai dengan
data sampel dan format file yang dimiliki, klik “OK”. Klik Save, semua kotak
dialog yang muncul pilih “OK” tunggu hingga proses selesai.
Apabila matrix tidak otomatis muncul, klik “Show Matrix” pada bagian kiri
bawah. Selanjutnya, pensejajaran sekuens DNA (alignment). Diklik “Matrix”,
kemudian dipilih “Align Multiple Sequences”, Dipilih “Opal” atau “ClustalW
Align” dan “Ok” untuk setiap kotak dialog yang muncul. Hasil yang didapat
kemudian dipotong guna menyamakan panjang dari sekuen yang akan dilakukan
analisis. Diklik nomor sekuen yang tidak rata, kemudian ditekan tombol ”Shift +
panah kiri” untuk memblok urutan basa pada bagian depan yang tidak rata,
kemudian ditekan tombol “Backspace” pada keyboard dan pilih “Yes”, sehingga
didapatkan sekuen DNA yang sejajar. Setelah selesai, hasil yang didapat
kemudian di export. Dengan cara diklik ”File” dipilih “export” dipilih format file
FASTA (DNA/RNA) dan “OK”. Dan akan muncul kotak dialong klik export.
Lalu ubah ekstensi file dari .nt menjadi .fas lalu klik “Save”.
Dibuka program MEGA, klik “File” laludipilih “Open A file/Session”. File
yang didapat dari langkah sebelumnya (file dalam bentuk .fas) lalu diklik “Open”
danakan muncul kotak dialog klik “Align”. Untuk mengetahui variasi basa
nukleotida dan conserved region dapat dilakukan Alignment pada toolbar, lalu
pilih dan klik ClustalW dan klik OK. Diklik OK tunggu sampai proses selesai,
dan muncul tampilan alignment variasi basa nukleotida sehingga dapat terlihat
daerah beda /mutasi serta daerah conserved region (ditandai dengan tanda“ * ”).
V. Hasil dan Deskripsi
Berdasarkan praktikum yang telah dilakukan, didapatkan hasil sebagai
berikut :
5.1. Analisis Mutasi
1 2 3
4 5
Keterangan:
1. Mutasi Delesi
2. Transversi
3. Transisi
4. Mutasi Substansi
5. Conserved Region
Deskripsi:
5.2. Jarak Genetik
1 2 3 4 5
1 Sismik 1 0.0050 0.0061 0.0071 0.0093
2 NR 148295.1 Pseudomonas 0.0127 0.0046 0.0051 0.0091
songnenensis strain NEAU-
ST5-5 16S ribosomal RNA
partial sequence
3 NR 169495.1 Pseudomonas 0,0190 0.0105 0.0050 0.0090
nitrititolerans strain GL 14
16S ribosomal RNA partial
sequence
4 NR 113617.1 Pseudomonas 0.0253 0.0127 0.0127 0.0088
oleovorans strain NBRC
13583 16S ribosomal RNA
partial sequence
5 NR 114167.1 Azotobacter 0.0443 0.0401 0.0380 0.0380
chroococcum strain NBRC
102613 16S ribosomal RNA
partial sequence
Deskripsi:
5.3. Pohon Filogenetik
Deskripsi:
Anda mungkin juga menyukai
- Isolasi DNADokumen24 halamanIsolasi DNAeka alishas100% (1)
- Najwa Maharani S - 08041282126043 - 4 SismikDokumen12 halamanNajwa Maharani S - 08041282126043 - 4 SismikNajwa SalsabilaBelum ada peringkat
- Cara Kerja Taksonomi Filogenetik SISMIK 2021Dokumen19 halamanCara Kerja Taksonomi Filogenetik SISMIK 2021Siti AisyahcantixBelum ada peringkat
- Tutorial Analisis Filogenetik Menggunakan Software MegaDokumen12 halamanTutorial Analisis Filogenetik Menggunakan Software MegaKepala LabBelum ada peringkat
- Sekuensing DNADokumen13 halamanSekuensing DNAAdhara ZephyraBelum ada peringkat
- 1.modul Praktikum BiosistematikaDokumen2 halaman1.modul Praktikum BiosistematikaReza FauziBelum ada peringkat
- Protokol Pelatihan BioinformatikaDokumen21 halamanProtokol Pelatihan BioinformatikaI EBelum ada peringkat
- Bioinformatika 4Dokumen9 halamanBioinformatika 4pwd bambangBelum ada peringkat
- C - 4 - 230110200159 - Hafidz Ar Rashif HabibiDokumen5 halamanC - 4 - 230110200159 - Hafidz Ar Rashif Habibi159Hafidz Ar Rashif HabibiBelum ada peringkat
- C1 - 08 - Pengenalan NCBIDokumen36 halamanC1 - 08 - Pengenalan NCBIAbel PutraBelum ada peringkat
- BIOINFORMATIKADokumen15 halamanBIOINFORMATIKAAsuler Weleh Weleh100% (3)
- Taksonomi Filogenetik (Molekuler)Dokumen11 halamanTaksonomi Filogenetik (Molekuler)silviyBelum ada peringkat
- Bioinfor 1Dokumen5 halamanBioinfor 1BENNY PRAMANA ZIHAD MARPAUNGBelum ada peringkat
- Bioinformatika FKH 2022Dokumen28 halamanBioinformatika FKH 2022Cecillia ParhusipBelum ada peringkat
- 13 Tugas Praktikum Hoirun NisaDokumen11 halaman13 Tugas Praktikum Hoirun NisaHoirun NisaBelum ada peringkat
- Panduan Praktikum Bioteknologi KelautanDokumen41 halamanPanduan Praktikum Bioteknologi KelautanRamadhani Marfatah100% (1)
- Kelompok 1 - Laporan Praktikum Sequencing - Biokimia - PSPB C 2019Dokumen6 halamanKelompok 1 - Laporan Praktikum Sequencing - Biokimia - PSPB C 2019Anggi Natasya100% (1)
- Tutorial Dan Praktek Interpretasi Data Hasil Sequence Secara Applied ComputerizeDokumen29 halamanTutorial Dan Praktek Interpretasi Data Hasil Sequence Secara Applied ComputerizeNova LRBelum ada peringkat
- Analisis Pohon FilogeniDokumen12 halamanAnalisis Pohon FilogeniSiti NovianaBelum ada peringkat
- Cara Menggunakan Aplikasi MEGA 5 MoleculDokumen5 halamanCara Menggunakan Aplikasi MEGA 5 MoleculAzizah MunitaBelum ada peringkat
- (Tasya Regita Maharani) Elektroforesis Sampel Untuk Deteksi Gen GAPDH Pada Sampel Jaringan Dan Instalasi Software Analisis QPCRDokumen12 halaman(Tasya Regita Maharani) Elektroforesis Sampel Untuk Deteksi Gen GAPDH Pada Sampel Jaringan Dan Instalasi Software Analisis QPCRTasya Regita MaharaniBelum ada peringkat
- Nelly Juenti - Resume 6 - Kelompok 2Dokumen9 halamanNelly Juenti - Resume 6 - Kelompok 2004Nelly JuentiBelum ada peringkat
- Acara 3Dokumen15 halamanAcara 3pricilia gitaBelum ada peringkat
- Modul-Sesi2 1Dokumen6 halamanModul-Sesi2 1Hilman TaufiqBelum ada peringkat
- Resume 6Dokumen9 halamanResume 6004Nelly JuentiBelum ada peringkat
- Laprak BlastDokumen11 halamanLaprak BlastintanBelum ada peringkat
- Modul Analisis Hasil SekuensingDokumen12 halamanModul Analisis Hasil Sekuensingunggarapunja12Belum ada peringkat
- Laporan P8 - Kelompok 4 Praktikum BiokimiaDokumen18 halamanLaporan P8 - Kelompok 4 Praktikum BiokimiaFirdaus ShofwanBelum ada peringkat
- Modul 6 MigDokumen6 halamanModul 6 MigPramadhana GemariantoBelum ada peringkat
- Analisis Dna Sequence Dengan Aplikasi MegaDokumen14 halamanAnalisis Dna Sequence Dengan Aplikasi Mega33. Ni Putu Ananda Yuliastri DewiBelum ada peringkat
- TrcytuiDokumen22 halamanTrcytuimaulidyaBelum ada peringkat
- Cara Kerja Mega 6Dokumen10 halamanCara Kerja Mega 6ita rianiBelum ada peringkat
- Laprak BLASTDokumen24 halamanLaprak BLASTMuhammadRezaPahlevi100% (2)
- Bioinformatika 4 - An Nissa Ade Astary Dewi - 119180020 - 2021Dokumen6 halamanBioinformatika 4 - An Nissa Ade Astary Dewi - 119180020 - 2021annissa 119180020Belum ada peringkat
- Kelompok 2 - P6Dokumen20 halamanKelompok 2 - P6irenBelum ada peringkat
- Laporan Praktikum Bioinformatika-DikonversiDokumen23 halamanLaporan Praktikum Bioinformatika-DikonversiNurlina Chindy Fatika Sari Nurlina Chindy Fatika SariBelum ada peringkat
- Membuat Pohon Filogenetik Citrus SP Menggunakan MEGA XIDokumen14 halamanMembuat Pohon Filogenetik Citrus SP Menggunakan MEGA XIIndah Nur SBelum ada peringkat
- Vina Safitri - Laporan Pratikum Analisis Data SequencingDokumen13 halamanVina Safitri - Laporan Pratikum Analisis Data SequencingNurul Ilma SeptianiBelum ada peringkat
- Analisis Pensejajaran Urutan DNA/protein Dengan Blast SystemDokumen20 halamanAnalisis Pensejajaran Urutan DNA/protein Dengan Blast SystemSiti Rahmaningsih100% (1)
- Laporan Praktikum 2 Ko KevDokumen18 halamanLaporan Praktikum 2 Ko KevMaria Angelina DjohanBelum ada peringkat
- Bab 3 Analisis GenomDokumen3 halamanBab 3 Analisis GenomMuhammad Arif SetiawanBelum ada peringkat
- Laporan Praktikum Pengantar BioinformatikaDokumen11 halamanLaporan Praktikum Pengantar BioinformatikaYusfida Mariatul HusnaBelum ada peringkat
- Buku Petunjuk Drylab Biosel 2010Dokumen14 halamanBuku Petunjuk Drylab Biosel 2010Putri Ayu SafitriBelum ada peringkat
- Jawaban EssayDokumen10 halamanJawaban EssayJeri Nobia PurnamaBelum ada peringkat
- Pengenalan NcbiDokumen35 halamanPengenalan Ncbitia100% (1)
- ArlequinDokumen5 halamanArlequinFiqitaBelum ada peringkat
- Modul NCBI Introduction BLAST 3D 10 AprilDokumen16 halamanModul NCBI Introduction BLAST 3D 10 AprilsarijuicyBelum ada peringkat
- Kelompok 6 Sekuensing Dan Analisis HomologiDokumen11 halamanKelompok 6 Sekuensing Dan Analisis Homologipanda biruBelum ada peringkat
- Aplikasi GenomikDokumen3 halamanAplikasi GenomikFitri Utami HasanBelum ada peringkat
- MAKALAH PbaDokumen15 halamanMAKALAH PbaFebi UtoyoBelum ada peringkat
- (Praktikan) Tugas Pendahuluan MODUL7Dokumen2 halaman(Praktikan) Tugas Pendahuluan MODUL7erfanus janeBelum ada peringkat
- Uts Bioinformatika Citra InnosensiaDokumen27 halamanUts Bioinformatika Citra InnosensiaCitra InnosensiaBelum ada peringkat
- Windi Nopitasari 19032162 Laporan Analisis Data SequencingDokumen13 halamanWindi Nopitasari 19032162 Laporan Analisis Data SequencingSiska TanjungBelum ada peringkat
- GENEBANK Adalah Database Nucleotida Yang KomprehensifDokumen2 halamanGENEBANK Adalah Database Nucleotida Yang KomprehensifTio UgantoroBelum ada peringkat
- Merancang ProbeDokumen11 halamanMerancang ProbeErlina Frizka0% (1)
- Hasil Diskusi Kelompok 5Dokumen3 halamanHasil Diskusi Kelompok 5dyah ayuBelum ada peringkat
- Praktikum Ii Pengenalan Situs BioinformaDokumen14 halamanPraktikum Ii Pengenalan Situs BioinformaAnkerz15Belum ada peringkat
- Modul Praktikum Isolasi DNA Buah Berdaging LunakDokumen4 halamanModul Praktikum Isolasi DNA Buah Berdaging LunakMeyga Evi Ferama Sari, S.Si, M.SiBelum ada peringkat
- Membuat Aplikasi Bisnis Menggunakan Visual Studio Lightswitch 2013Dari EverandMembuat Aplikasi Bisnis Menggunakan Visual Studio Lightswitch 2013Penilaian: 3.5 dari 5 bintang3.5/5 (7)
- BAB 4 KL SiswanDokumen2 halamanBAB 4 KL Siswannur aisyah adzutiBelum ada peringkat
- Hasil SismikDokumen10 halamanHasil Sismiknur aisyah adzutiBelum ada peringkat
- Caker Acara 13Dokumen2 halamanCaker Acara 13nur aisyah adzutiBelum ada peringkat
- Tabel Hasil Identifikasi Tekstur TanahDokumen1 halamanTabel Hasil Identifikasi Tekstur Tanahnur aisyah adzutiBelum ada peringkat
- LAMPIRAn GenetDokumen1 halamanLAMPIRAn Genetnur aisyah adzutiBelum ada peringkat
- Cek Plagiat 1Dokumen3 halamanCek Plagiat 1nur aisyah adzutiBelum ada peringkat
- Cek Plagiat 1Dokumen3 halamanCek Plagiat 1nur aisyah adzutiBelum ada peringkat
- DapussssDokumen1 halamanDapussssnur aisyah adzutiBelum ada peringkat
- Daftar PustakaDokumen9 halamanDaftar PustakaDo AdzutiBelum ada peringkat
- Bab 45 BungaDokumen4 halamanBab 45 Bunganur aisyah adzutiBelum ada peringkat
- Caker Acara 11Dokumen2 halamanCaker Acara 11nur aisyah adzutiBelum ada peringkat
- Rajinbacaebook - Pramoedya - Bumi Manusia PDFDokumen314 halamanRajinbacaebook - Pramoedya - Bumi Manusia PDFTobing89% (9)
- Caker Acara 7Dokumen1 halamanCaker Acara 7nur aisyah adzutiBelum ada peringkat
- Caker Acara 8Dokumen1 halamanCaker Acara 8nur aisyah adzutiBelum ada peringkat
- Remidi B. IndoDokumen5 halamanRemidi B. Indonur aisyah adzutiBelum ada peringkat
- Bab 5 HasilDokumen3 halamanBab 5 Hasilnur aisyah adzutiBelum ada peringkat
- Hasil Acara 8Dokumen2 halamanHasil Acara 8nur aisyah adzutiBelum ada peringkat
- Manlab BiolDokumen4 halamanManlab Biolnur aisyah adzutiBelum ada peringkat
- Hasil Acara 7Dokumen1 halamanHasil Acara 7nur aisyah adzutiBelum ada peringkat
- Atom Karbon Mempunyai Ke KhasanDokumen25 halamanAtom Karbon Mempunyai Ke Khasannur aisyah adzutiBelum ada peringkat